G-kvadruplex
A G-kvadruplex másodlagos szerkezetek (G4) guaninban gazdag szekvenciák által jönnek létre.[2] Ezek helikálisak és 1,[3] 2[4] vagy 4 szálas guanintetrádokból állnak.[5] Az egymolekulás változatok gyakoriak a kromoszómák végén lévő telomereken és számos gén transzkripciószabályzó részein mikrobákban[6][7] és gerincesekben,[8][7] beleértve humán onkogéneket.[9] 4 guanin Hoogsteen-hidrogénkötéssel négyzetes szerkezetű guanintetrádot alkothat, és 2 vagy több guanintetrád (folyamatos G-sorozatokból) egymáson G-kvadruplexet alkot.
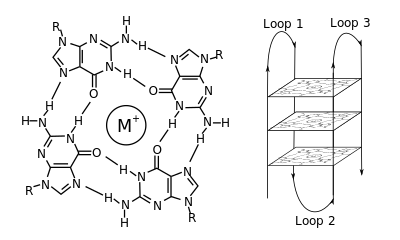
A G-kvadruplexek elhelyezkedése és kötései nem véletlenszerűek, és különös funkcióik vannak. A kvadruplexet egy tetrádpárok közt középre kerülő kation, különösen a kálium jelenléte stabilizálja.[3] Alkothatja DNS, RNS, ZNS vagy PNS, és lehet molekulán belüli, két- vagy négymolekulás.[10] A szálak vagy azok részeinek irányától függően a szerkezetek lehetnek párhuzamosak vagy antipárhuzamosak. A G-kvadruplexek DNS- vagy RNS-szekvenciamotívumokból előrejelezhetők,[11][12] de valódi szerkezetük változhat motívumon belül és motívumok közt, amelyekből genomonként több mint 100 000 is lehet. Alapvető genetikai folyamatokban való részvételük miatt kutatják a telomerek, a génszabályzás és a funkciós genomika területén.[13][14]
Története
szerkesztésA nagy guaninasszociációs szerkezeteket 1962-ben azonosították guaninasszociált gélszerű anyagokon keresztül.[15] E kutatás határozta meg a 4 szálú DNS-szerkezetek magas guaninasszociációval való összefüggését, melyeket 1987-ben eukarióta telomerekben fedeztek fel.[16] A G-kvadruplex felfedezésének fontosságát a következő idézet mutatta meg:
Ha in vitro keletkeznek G-kvadruplexek, a természet alkotott egy módot azok in vivo használatára”
A G-kvadruplexek in vivo funkciójának kutatása a nagyméretű genomszintű analízis hatására megnőtt, melyben a potenciális G-kvadruplex-alkotó (pG4) szekvenciák nagy gyakoriságát mutatták ki humán, csimpánz- egér- és patkánypromoterekben, melyeket a 2007. áprilisi első nemzetközi G-kvadruplex-találkozón mutattak be a kentuckyi Louisville-ben.[7] 2006-ban néhány bakteriális promoterben is kimutattak G-kvadruplexeket, G-kvadruplex-mediált génszabályzást feltételezve.[6] A G-kvadruplexek in vivo gyakorisága révén e szerkezetek biológiailag fontosak az onkogének promotereire és a telomerekre való hatásuk révén. Jelenleg a kutatás a G-kvadruplexek funkcióinak azonosítására bizonyos onkogéneknél, valamint a G-kvadruplexeken alapuló hatékony kezelések felfedezésére irányul. A G-kvadruplexek korai bizonyítéka sejtekből való izolációjuk volt,[17] később annak megfigyelésével is igazolták, hogy egyes DNS-helikázok ilyen szerkezetekhez közel vannak nagy mennyiségben.[18]
Topológia
szerkesztésA tetrádképzésben részt vevő nukleinsavak gatározzák meg a kvadruplex szerkezetét. A rövid, egyetlen 3 vagy több guainból álló sorozatból álló szekvenciák 4 önálló szálat igényelnek a kvadruplexhez, vagyis az ilyen kvadruplex tetramolekulás. A G4 DNS eredetileg az ilyen szerkezetekre utalt, melyek a meiózisban lehetnek fontosak.[5] Azonban a molekuláris biológia modern szóhasználatában a G4 bármilyen molekularitású kvadruplexet jelenthet. A hosszabb, két folytonos 3 vagy több guaninból álló sorozatot tartalmazó kvadruplexekhez 2 ilyen szekvencia is elég. Ezek a kvadruplexek így bimolekulárisak. Végül a 4 különböző guaninsorozatot tartalmazó szerkezetek önmaguktól képesek stabil kvadruplexet alkotni, így intramolekulárisak.[19]
A guaninbázisok elrendezésétől függően egy kvadruplex számos alakot felvehet különböző konfigurációkkal.[20] Ha minden DNS-szál azonos irányba halad, a kvadruplex párhuzamos. Intramolekuláris kvadruplexek esetén ekkor minden körnek propellernek kell lennie, melyek a kvadruplex oldalain vannak. Ha egy vagy több guaninsorozat 5’-3’ iránya a többivel ellentétes, a kvadruplex antipárhuzamos. Ekkor a guaninsorozatokat egymáshoz csatlakoztató körök az intramolekulás kvadruplexekben vagy átlósak (ellentétes guaninsorozatokat csatlakoztatók), vagy laterálisak (oldal menti), így két szomszédos guaninsorozatot csatlakoztatnak.
A kétszálú DNS-ből alkotott G-kvadruplexekben lehetségesek szálközi topológiák is.[21][22] E kvadruplexek mindkét DNS-szálból tartalmaznak guanint.
Szerkezete és szerepe a genomban
szerkesztésA humán genom szekvenálása után számos kvadruplexalkotásra képes guaningazdag szekvenciát találtak.[23] A sejt típusától és sejtciklusban való állapotától függően a közvetítő tényezők, például a kromatin DNS-kötő fehérjéi és más környezeti tényezők befolyásolhatják a kvadruplexkeletkezést. Például a molekulazsúfoltság kvantitatív termodinamikai elemzése alapján az antipárhuzamos G-kvadruplexet a molekulazsúfoltság stabilizálja.[24] Ez feltehetően a DNS-hidratáció megváltozása és ennek Hoogsteen-bázispárkötésre gyakorolt hatása okozza.[25] Ezek gyakran kromoszómák végén fordulnak elő. Továbbá a G-kvadruplex-képzés valószínűsége transzkripció során RNS-ben hajtű- vagy G-kvadruplex-szekvenciák létrehozásához a hajtűlétrehozó szekvencia helyétől függ.[26]
Mivel a javító enzimek a lineáris kromoszómák végeit sérült DNS-ként észlelnék, és ezeket káros hatásként dolgoznák fel, egyértelmű jelzés és szigorú szabályzás kell a kromoszómák végein. A telomerek lehetővé teszik e jelzést. Ezek guaninban gazdagok, hajlamosak G-kvadruplex-képzésre, gyakran a kromoszómák végén vannak, és segítik a genom integritásának megőrzését a végek instabilitástól való védelmével.
E telomereket hosszú CCCTAA:TTAGGG ismétlődések jellemzik. Az ismétlődéek 3'-kiemelkedéssel érnek véget 10-50 egyszálú TTAGGG ismétlődéssel. A heterodimer-komplex ribonukleoprotein telomeráz a DNS-szálak 3'-végéhez TTAGGG sorozatokat ad. A G-gazdag meghosszabbítás képes másodlagos szerkezetek, például G-kvadruplexek létrehozására, ha hosszabb 4 TTAGGG ismétlődésnél. Ezek jelenléte megakadályozza a telomer telomeráz általi elongációját.[27]
Előfordulás
szerkesztésTelomerkvadruplexek
szerkesztésA telomerismétlődésekről számos élőlényben kimutatták in vitro a kvadruplexek létrehozását, később pedig kimutatták ugyanezt in vivo.[28][29] A humán telomerismétlődés (mely minden gerincesnél ugyanaz) a TTAGGG számos ismétlődéséből áll, és az így keletkező kvadruplexek 5-8 nm-es gyöngyszerű szerkezetekben fordulhatnak elő, és NMR-, TEM- és röntgenvizsgálatokkal meghatározták.[30] E kvadruplexek keletkezése csökkenti a telomeráz aktivitását, mely a telomerek hosszáért felel, és a rákok 85%-áért felel. Ez egy aktív kutatási célpont gyógyszerek, például a telomesztatin számára.
A G-kvadruplex-mediált genominstabilitás sok rákkal összefüggő egypontos nukleotidvariánst okoz.[31]
Telomeren kívüli kvadruplexek
szerkesztésA kvadruplexek nemcsak telomerekben vannak jelen. Humán, csimpánz-, egér- és patkánygenomok elemzése számos pG4 szekvenciát mutatott a telomereken kívül. Sok ilyen kvadruplex volt promoterekben, és ezek a fajok közt állandók maradtak.[6][7] Sok G-kvadruplex található az E. coliban és több száz más mikrobiális genomban. Itt is a gerincesekhez hasonlóan a G-kvadruplexek gyakoriak a promoterekben.[6] Ezenkívül több mint egymilliárd éves G-kvadruplex-lokusz is ismert növényekben és algákban az RNS-polimeráz II nagy alegységének génjében.[32] Bár e tanulmányok G-kvadruplex-mediált génszabályzást jeleztek előre, nem valószínű, hogy minden pG4 in vivo is keletkezik. A c-myc protoonkogén egy génaktivitásért felelős nukleáz-hiperszenzitív régióban G-kvadruplexet alkot.[33][34] További, promoterében G-kvadruplexet tartalmazó gének például a tyúk-β-globin génje, a humán ubikvitin-ligáz RFP2, valamint a c-kit, bcl-2, VEGF, a H-ras és az N-ras.[35][36][37]
Kvadruplexkeletkezési szabályon alapuló genomfelméréseket is végeztek 276 000 humán feltételezett kvadruplexszekvencián, de ezek nem mindegyike alakít in vivo kvadruplexet.[38] Hasonló tanulmányok azonosítottak feltételezett G-kvadruplexeket prokariótákban, például E. coliban.[39] Számos lehetséges modell van a kvadruplexek génaktivitás-módosítására. Egy modell látható lejjebb, ahol egy promoterben vagy ahhoz közel G-kvadruplex alakul ki, blokkolva a gén transzkripcióját, inaktívvá téve azt. Egy másik modellben a nem kódoló DNS-ben lévő kvadruplex lehetővé teszi a kódoló DNS nyitott állását és a megfelelő gén kifejeződésének növelését.
Funkció
szerkesztésFeltehetően a kvadruplexképződés az immunglobulin-nehézlánc-váltásban játszik szerepet.[5] A sejtek a kvadruplexek feloldására fejlesztettek módszereket, mivel a kvadruplexképződés káros lehet egy sejtnek, így a WRN helikáz és a Bloom-szindróma-fehérje nagy affinitással old fel G-kvadruplexeket.[40] A DEAH/RNS-helikáz DHX36 fontos G-kvadruplex-reszolváz.[41][42] 2009-ben egy áttétfehérjéről, az NM23H2-ről (más néven NME2) kiderült, hogy a c-myc promoterének kvadrulpexével közvetlen kölcsönhatásba lép, és transzkripcióját szabályozza.[43][44] 2017-ben kiderült, hogy az NM23H2 a humán telomeráz hTERT promoterének G-kvadruplexével is kölcsönhatásba lép, annak expresszióját szabályozza.[45] 2019-ben a 2. telomerkötő faktorról (TRF2, TERF2) kiderült, hogy több ezer telomeren kívüli G-kvadruplexhez kötődik a TRF2 ChIP-seq. révén[46] Számos tanulmány ír a kvadruplexek pozitív és negatív transzkripciószabályzásáról, beleértve a gének, például a hTERT epigenetikai szabályzását.[45] A G-kvadruplexekről kiderült az is, hogy az immunglobulin nehéz génjeinek és a patogén Neisseria pilinantigén-variációs rendszerének programozott rekombinációjáról.[47] A kvadruplex szerepei nem jól ismertek. A humán sejten belüli G-kvadruplexek közvetlen megjelenítése[48] és egy G-kvadruplexhez kötött RNS-helikáz társkristályszerkezete[49] fontos információt adott a sejtbiológiai fontosságukról. A kvadruplexek lehetséges pozitív és negatív telomerreplikációs szerepei és funkciói ismeretlenek. A T-hurkok és a G-kvadruplexek a DNS két olyan szerkezete, melyek megvédik a telomervégeket és szabályozzák a telomerhosszt.[50]
Genomszabályzás G-kvadruplexek kialakulásával
szerkesztésSzámos genomszabályzó folyamat kapcsolatban állhat G-kvadruplexek keletkezésével, mivel a purin- vagy pirimidinmentes (AP-helyek) javításában játszhat szerepet.[51] Az AP helyek hozzárendelésének új módszerét (AP-seq) 2020-ban fejlesztette ki A. R. Poetsch, és biotinnal jelölt aldehidreaktív vizsgálatot (ABP) használ a genom azon helyeinek jelölésére, ahol az AP-helyek jelentős károsodást okoztak.[52] Egy másik genomszekvenálási módszer, a ChIP-szekvenálás használható mind az AP-helyek károsodásának, mind az azt javító AP-endonukleáz 1 (APE1) elemzésére. Mind a ChIP-szekvenálás, mind az ABP alapján az AP-helyek sérülése nem véletlenszerű, és gyakoribb a genom egyes promotert és kifejeződést növelő részeiben, melyek egy része kapcsolatban állhat a tüdő-adenokarcinómával és a vastagbélrákkal.[53] Az AP-károsodás főként a genom PQS-részeiben fordul elő, ahol a G-kvadruplex-keletkezés szabályozott, és a báziseltávolító javítás ezt növeli.[53] A báziseltávolító javítás mértéke az öregedéssel csökken, mivel az azt lehetővé tevő részek bomlanak, ami számos betegség, például az Alzheimer-kór kialakulásához vezethet.[54] E G-kvadruplexeket feltehetően a szuperhelicitás okozza a promoterekben, mely a kettős hélix felbomlását segíti, és G-kvadruplexet kialakítva kapcsolja a szálakat a guaningazdag részeken.[55] A BER-út jelez oxidatív báziskárosodásnál, ahol a 8-oxoguanin-DNS-glikoziláz 1-hez (OGG1), APE1-hez és a G-kvadruplexhez hasonló szerkezetek nagy szerepet játszanak a javításban Ezen enzimek vesznek részt a BER-ben egyes DNS-léziók, például a 7,8-dihidro-8-oxoguanin (8-oxoG) javításában, mely a guaninból keletkezik oxidatív stressz hatására.[56]
Az endogén oxidációs DNS-károsodás szerepe a G4-kialakulásban
szerkesztésA G-kvadruplex guaninjáé a legalacsonyabb elektronpotenciál, így hajlamosabb 8-oxoguaninná alakulásra, mely endogén oxidált DNS-bázis. Mivel a guanin elektronpotenciálja alacsonyabb a többi nukleobázisénál,[57] a 8-Oxo-2′-dezoxiguanozin (8-oxo-dG) a DNS-oxidáció gyakori terméke. Koncentrációja a sejtre ható oxidatív stressz mértékét mutatja.[58] A DNS oxidatív károsodásakor a guanin egy lehetséges szerkezeti változásakor enol forma jelenik meg, a 8-OH-G. Ezen oxidatív termék az eredeti sérült guanin, a 8-oxo-G tautomériája révén keletkezik, és a DNS szerkezeti károsodását mutatja meg. E forma lehetővé teszi a báziseltávolításos javítást (BER) végző OGG1 enzimnek a 8-OH-G APE1 segítségével való eltávolítását, AP-helyet létrehozva.[56][54] Az AP-helyek azon helyek a DNS-ben, ahol nincs purin- vagy pirimidinbázis a DNS-sérülés miatt, ez a leggyakoribb endogén DNS-károsodás a sejtekben. Ezek spontán vagy a módosult bázisok (például a 8-OH-G) eltávolításakor keletkezhetnek.[52] Az AP-hely létrejötte lehetővé teszi a duplex DNS olvadását a PQS megmutatásához,[54] G-kvadruplexet létrehozva. A genomszintű ChIP-szekvenálás, a sejtalapú assay-k és az in vitro biokémiai elemzések révén összeköthetők az oxidáltbázis-alapú AP-helyek és a G-kvadruplexek keletkezése.[53]
A DNS oxidációjának kapcsolata betegségekkel
szerkesztésEzenkívül a 8-oxo-dG koncentrációja a sejtre ható oxidatív stressz biomarkere, és a nagy mennyiségű oxidatív stressz összefügg a karcinogenezissel és más betegségekkel.[59] A 8-oxo-dG keletkezésekor inaktiválhatja az OGG1-et, megakadályozva a guaninoxidáció okozta DNS-károsodást.[53] Ez lehetővé teszi a javítatlan károsodások növekedését nem osztódó sejtekben, például az izomban, és öregedést is okozhat.[58] Továbbá az oxidatív DNS-károsodás a karcinogenezishez génexpresszió-modulációval vagy mutációokozással járul.[58] Ha a 8-oxo-dG-t a BER javítja, a javítófehérje egy része ott marad, mely epigenetikai változásokat vagy a génexpresszió modulációját okozhatja.[60] A humán timidin-kináz-génbe kerülő 8-oxo-dG-ről kiderült, hogy ha változatlanul marad, és nem javítja ki a BER, gyakori mutációkat és végül karcinogenezist okozhat.[53][54]
Az APE1 szerepe a génszabályzásban
szerkesztésAz AP-endonukleáz 1 (APE1) a G-kvadruplexek kialakulásáért és erősítéséért felelős enzim. Fő célja az AP-helyek javítása BER-rel. Az APE1 nagyon fontos, mivel az AP-helyek okozta sérülés a leggyakoribb endogén DNS-sérülés.[60] Egyes purinbázisok, például a guanin oxidációja oxidált nukleotidokat hoz létre, melyek a DNS-funkciót a szekvenciában lévő hibás nukleotidokkal gátolja.[58] Ez gyakoribb az oxidált szerkezeteket, például 8-oxoguanint alkotó PQS-szekvenciákban. Amint a sejt észleli az oxidatív stresszt és a károsodást, a helyszínre küld OGG1-et, melynek fő funkciója a BER útvonal elindítása.[53] Ezt az OGG1 az oxidált bázis eltávolításával hajtja végre, AP-helyet létrehozva, elsősorban negatív szuperhelicitással.[55] Ez jelet küld APE1-kötéshez, mely a nyitott duplexhez kötődik.[59] Az APE1-kötés stabilizálja e helyen a G-kvadruplexek keletkezését. Ez a G-kvadruplexek keletkezését hajtással segíti.[61] E folyamat 4 bázist helyez egymáshoz közel, melyeket Hoogsteen-bázispárok tartanak össze. Ezután az APE1-et több lizin acetilezi a kromatinon, acetil-APE1-et (AcAPE1) létrehozva. Ez fontos a BER útvonalhoz, mivel transzkripciós koaktivátorként vagy korepresszorként működik, transzkripciós faktorokat helyezve a károsodás helyére, lehetővé téve a génexpresszió szabályzását.[62] Az AcAPE1 továbbá lehetővé teszi, hogy az APE1 tovább kapcsolódjék a disszociáció késleltetésével, lehetővé téve a javítás hatékonyságának növelését.[63] Az AcAPE1 deacetilezése helyezi el a TF-eket, ahol az APE1 disszociál a G-kvadruplexekről.[64]Az APE1 és AcAPE1 mennyiségének csökkenésekor a G-kvadruplexek keletkezése is csökkent, ami alapján az APE1 fontos e szerkezetek keletkezéséhez. Azonban nem minden G-kvadruplexhez kell APE1, egyes esetekben nagyobb G-kvadruplexek keletkeztek APE1 nélkül.[53] Tehát az APE1-nek két fontos feladata van: a G-kvadruplexek stabilizálása és a transzkripciós faktorok AP-helyre helyezése.
Rák
szerkesztésTelomerek
szerkesztésA G-kvadruplex-képző szekvenciák gyakoriak az eukarióta sejtekben, különösen a telomerekben, az 5'-nem transzlált szálakon és a transzlokációs forrópontokon. A G-kvadruplexek gátolhatják a normál sejtfunkciót, és az egészséges sejtekben könnyen szétválasztja a helikáz. Azonban a mutált helikázú ráksejtekben e komplexek nem válnak szét, ami a sejt károsodásához vezethet. Ez károsodott rákos sejtek replikációját okozza. A G-kvadruplexek stabilizálása gátolja a sejtnövekedést és -replikációt, sejthalálhoz vezetve.[65]
Promoterek
szerkesztésA G-kvadruplexek asszociációja mellett egyes protoonkogén-promoterek mellett is találhatók G-kvadruplexek. Ezek általában párhuzamos szálú G-kvadruplexek.[66] Ilyen onkogének például a c-KIT, a PDGF-A, a c-Myc és a VEGF, ami e másodlagos szerkezet fontosságát mutatja a rák növekedésében és fejlődésében. Míg a G-kvadruplex kialakulása valamennyire eltér a különböző onkogének promoterei közt, ezek stabilizációja észlelhető a rák fejlődése során.[67] A terápiás kutatások a G-kvadruplex stabilizációját célozzák a szabályozatlan sejtnövekedés és -osztódás megállítására.
Egy különösen fontos génrész, a c-myc út fontos a fehérjetermék c-Myc szabályzásában. E termékkel a c-Myc az apoptózis, a sejtnövekedés, -fejlődés és a humán telomeráz-reverztranszkriptáz transzkripciójának irányításában vesz részt.[68] A c-Myc-promoter G-kvadruplexének NM23H2-vel való kölcsönhatása szabályozza a ráksejtek c-Myc fehérjéjét.[43]
A c-myc hTERT általi szabályzását a promoter G-kvadruplexe közvetlenül szabályozza az NM23H2 faktorral való kölcsönhatással, és az epigenetikai módosulások az NM23H2-től függnek.[45] 2020-ban a hTERT epigenetikai szabályzásáról kiderült, hogy a hTERT-promoter G-kvadruplexétől függ a TRF2 telomerfaktor révén.[69]
Egy másik génútvonal a VEGF génen alapul, mely az angiogenezisben játszik szerepet. Az intramolekuláris G-kvadruplex keletkezését a VEGF gén promoterének polipurin szakaszával mutatták ki. A G-kvadruplex in vivo funkciójáról szóló 2005-ös tanulmányok szerint a G-kvadruplex stabilizációja irányítja a VEGF transzkripcióját az útvonal TF-einek gátlásával. Az intramolekulás G-kvadruplexek nagyrészt ezen útvonal guaningazdag promoterrészében találhatók.[70] A ciklindependens sejtciklusellenőrzőpontkináz-inhibitor CDKN1A (más néven p21) génjének promoterében van G-kvadruplex. Ennek TRF2-vel való kölcsönhatása a p21 epigenetikai szabályzását okozta, melyet 360A G-kvadruplex-kötő ligandummal ellenőriztek.[71]
A hipoxiaindkálható faktor 1ɑ (HIF-1ɑ) a hipoxia-válaszelemhez (HRE) kapcsolódva fontos a rákjelzésben hipoxia esetén, elkezdve az angiogenezist. 2005-ös tanulmányok révén megismerhetővé vált e génútvonal, a polipurin- és polipirimidin részek lehetővé teszik ennek transzkripcióját és az intramolekulárif G-kvadruplex keletkezését. Azonban nem ismert, hogy a G-kvadruplex keletkezése pozitívan vagy negatívan befolyásolja a gén expresszióját.[72]
A c-kit onkogén RTK-t kódoló útvonalt használ, melynek expressziós szintjei bizonyos ráktípusokban nagyobb. A guaningazdag szekvenciájáról kiderült, hogy képes számos kvadruplex képzésére, de még nem ismert ennek hatása a c-kit-útvonalra, de e kvadruplex számos fajban előfordul.[37]
A RET onkogén egyes rákokban gyakori kináz transzkripcióját célozza. Ennek promoterének guaningazdag része szükségtelenné teszi a tirozin-kináz alapvető transzkripcióját. Egyes ráktípusokban a RET expressziója nagyobb. Ezen útvonal vizsgálata alapján a promoterben G-kvadruplex alakulhat ki, és ez megfelelő terápiás célpont.[73]
Egy PDGF-A-t (vérlemezke-alapú növekedési faktor) használó onkogénútvonal a sebgyógyulást használja, és mitogén növekedési faktorként működik. A PDGF nagy mértékű expressziója erősebb sejtnövekedéssel és rákkal függ össze. Promoterében a guaningazdag szakasz képes párhuzamos G-kvadruplexeket alkotni, és feltehetően a PDGF-A transzkripciós szabályzásában játszik szerepet. A G-kvadruplexek jelenlétét a TMPyP4 és a promoter kölcsönhatása révén fedezték fel.[74]
Terápia
szerkesztésA telomerek általában G-kvadruplexekből állnak, és a terápiakutatás és -felfedezések fontos célpontjai. E komplexek nagy affinitást mutatnak a porfingyűrűkre, ami hatékony rákellenes szerré teszi őket. Azonban a TMPyP4 használata korlátozott, mivel nem rákszelektív a ráksejttelomerek és és a normál dsDNS felé. Ennek megoldására 5-metilszármazékát is előállították, mely csak a G-kvadruplex-DNS-t célozza, jobban korlátozva a sejtnövekedést a TMPyP4-nél.[75]
A ligandumtervezés és -fejlesztés továbbra is fontos kutatási terület a G-kvadruplexek mennyisége és konformációs különbségei miatt. Egy kvindolintartalmú ligandum, a SYUIQ-05 a G-kvadruplexek stabilizációját használja a c-Myc fehérje és a humán telomeráz-reverztranszkriptáz keletkezésének gátlásához. E fő útvonal e rész célzására telomerázelongáció-hiányt okoz, leállítva a sejtfejlődést. Ezen kívül további kutatások kellenek egygénes célpont megtalálásához a nem kívánt reakciók csökkentéséért és a tumorellenes hatás növeléséhez.[68]
Kvadruplexkötő ligandumok
szerkesztésA G-kvadruplexek keletkezésének indukálására vagy azok stabilizálására G-kvadruplex-kötő anyag használható. Számos ligandum, melyek lehetnek kis molekulák és fehérjék is, kapcsolódhatnak a G-kvadruplexhez. E ligandumok lehetnek természetesek vagy szintetikusak.
A kationos porfinok és a telomesztatin be tudnak ágyazódni G-kvadruplexek közé.
A ligandumok kapcsolódása G-kvadruplexekhez fontos a rákellenes szereknél, mivel a G-kvadruplexek jellemzően transzlokációs forrópontokon vannak. Az MM41, egy szelektíven a BCL-2 promoteren lévő kvadruplexhez kapcsolódó ligandum központi magból és 4 oldalláncból áll. A ligandum alakja fontos, mivel az egymásra épülő kvartettekből és az ezeket összetartó nukleinsav gyűrűkből állnak. Amikor ehhez kötődik, az MM41 központi kromoforja a 3'-terminális G-kvartetten van, a ligandum oldalláncai a kvadruplexek gyűrűihez kapcsolódnak. A kromofor és a kvartett π–π kötéssel kapcsolódnak egymáshoz, míg az oldalláncok és a gyűrűk nem kapcsolódnak egymáshoz, de közel vannak. A gyűrűk helyzetének változékonysága teszi a kötést erőssé.[76]
Egy kationos porfin, a TMPyP4 ismert G4-kötő és MYC-represszióra képes ligandum. A TMPyP4 kötésének módja hasonlít az MM41-hez: a gyűrű a külső G-kvartetthez kapcsolódik, az oldalláncok a G4-gyűrűkhöz asszociálnak.[77]
A G-kvadruplexekhez kötődni képes ligandumok tervezésekor a ligandumok affinitása párhuzamos G-kvadruplexekhez nagyobb. A kisebb oldalláncú ligandumok jobban kötnek a kvadruplexhez, mivel a kisebb ligandumok elektronsűrűsége nagyobb. Továbbá a ligandumok kisebb oldallánchoz kisebb, erősebb kötéseket létesítenek. A mozgó oldalláncú ligandumok erősebben asszociálnak G-kvadruplexekhez, mivel a G4-gyűrű és a ligandumoldalláncok konformációja igazodhat.[78]
Kvadruplex-előrejelzési módszerek
szerkesztésA kvadruplexképzésre képes szekvenciák azonosítása fontos a szerepük megértésében. Általában egyszerű mintaegyezést használnak erre a szálon belüli kvadruplex képzésére alkalmas szekvenciákra: d(G3+N1-7G3+N1-7G3+N1-7G3+), ahol N tetszőleges nukleobázis (beleértve a guanint).[79] E szabályt gyakran használják online algoritmusok. Bár e szabály hatékonyan azonosít G-kvadruplex-képző helyeket, azonosíthat ezenkívül triplex-[80] és i-motívumú C-szál-képzésre alkalmas nem tökéletes homopurintükör-ismétlődések egy részét is.[81] Továbbá e szekvenciák képesek csúszott és visszahajtott szerkezetek azonosítására is, melyek kvadruplex-[4] és triplex-DNS képzésére alkalmasak.[82] Egy tanulmányban,[83] S. S. Smith kimutatta, hogy e motívumok bázispáronkénti megfigyelt száma (frekvenciája) a teljes elérhető genomszekvenciával rendelkező Eumetazoa-fajokban nagyobb. Ez alapján e szekvenciák pozitív szelekció alatt lehetnek, melyet a nem B-szerkezet kialakulását megakadályozó rendszerek fejlődése tett lehetővé.
2016-tól fejlett webalapú eszköztárak érhetők el G-kvadruplex-képző szekvenciák azonosítására, például a G4Hunter nyílt hozzáférésű felhasználóbarát, csúszóablakos változata[84] vagy a gépi tanuláson alapuló G4RNA Screener.[85]
Módszerek G-kvadruplexek tanulmányozására
szerkesztésSzámos kísérleti módszert fejlesztettek ki G-kvadruplexek azonosítására. Ezek két nagy csoportba (biofizikai és biokémiai) sorolhatók.[86]
Biokémiai
szerkesztésA G-kvadruplex-képződés hosszabb szekvenciában való vizsgálatára biokémiai módszerek használhatók. A DNS-polimeráz stop-assay-ben a G-kvadruplex DNS-templátban való keletkezése a polimeráz megállását okozó akadály lehet, mely a primer meghosszabbítását megállítja.[87] A dimetil-szulfát (DMS)-piperidin bontóassay azon alaul, hogy a G-kvadruplex képződése megakadályozza a DMS általi guaninmetilációt, vvédelmi mintához vezetve a DNS G-kvadruplexénél a piperidines bontás után.[88]
Biofizikai
szerkesztésA G-kvadruplex alakja meghatározható a körkörös dikroizmus jeleinek vizsgálatával adott hullámhosszokon.[89] A párhuzamos G-kvadruplexek 240 nm-es CD-jele negatív, 262 nm-es jele pozitív, az antipárhuzamosak jele 262 nm-en negatív, 295-ön pozitív. A G-kvadruplex-képződés igazolásához a CD-kísérlet nem G-kvadruplex-stabilizáló (Li+), illetve G-kvadruplex-stabilizáló (K+ vagy G-kvadruplex ligandumokkal) és a távoli UV tartományban (180-230 nm) kell vizsgáni. Ehhez hasonlóan a G-kvadruplexek hőstabilitása az UV-jel 295 nm-en való vizsgálatával azonosítható.[90] A G-kvadruplexek olvadásakor az UV-elnyelés 295 nm-en csökken, a G-kvadruplexre jellemző hipokrómos váltást okozva. További megközelítés a G-kvadruplex észlelésére például a nanopórus-alapú módszer. Először kiderült, hogy a biológiai nanopórusok képesek a G-kvadruplexek észlelésére méretalapú kizárás alapján és a G-kvadruplexek és a fehérjék nanoüregei közti specifikus interakciókkal.[91] Egy újabb megközeítésbem a szilárdtest-nanopórusok és a DNS-nanotechnológia használatosak a G-kvadruplexek észleléséhez dsDNS-ben és a G-kvadruplex-keletkezés észlelésére.[92]
Szerepe neurológiai betegségekben
szerkesztésA G-kvadruplexek kétféleképp játszhatnak szerepet neurológiai betegségekben. Az egyik mód a G-ismétlődések nyújtása génekben, melyek G-kvadruplex-képződést okoznak, melyek közvetlenül okozzák a betegséget, például a C9orf72 gén és az amiotrófiás laterálszklerózis (ALS) vagy a frontotemporális demencia (FTD) esetén. A másik mód a mutációk, melyek megváltoztatják a G4-kötő fehérjéket, például az 1. törékeny X-gén és a törékeny X-szindróma esetén.[93]
A C9orf72 a C9orf72 fehérjét kódolja, mely az agyban a neuronok citoplazmája és a preszinaptikus végeken található.[94] Mutációi összefüggnek az FTD és az ALS kialakulásával.[95] E két betegség a GGGGCC (G4C2) ismétlődésekkel függ össze az 1. intronjában. Általában 2-8 G4C2 ismétlődés van, de FTD és ALS esetén ez 500-tól néhány ezerig terjedhet.[96][97] Az ebből átírt RNS stabil G-kvadruplexeket alkot, és a DNS G4C2-ismétlődései vegyes párhuzamos-antipárhuzamos G-kvadruplexeket képesek alkotni.[98][99] Ezen G4C2-tartalmú átiratok kapcsolódhatnak egymáshoz, és sok fehérjét, például nukleolint is elválaszthatnak. A nukleolin a riboszóma-szintézisben és -érésben fontos, és a nukleolin mutált RNS-átirattal való elválasztása akadályozza a nukleólusz funkcióját és az rRNS-szintézist.[100]
A törékeny X-retardatio-fehérje (FMRP) sokszor expresszált fehérje, melyet az FMR1 gén kódol, és G-kvadruplexekhez kötődik, és a szinaptikus plaszticitásban fontos.[101] Az FMRP negatív transzlációszabályzó, kötése stabilizálja a G-kvadruplexeket az mRNS-ben, gátolva a riboszómaelongációt a neuron dendritjeiben, és irányítva az átirat kifejezésének idejét.[102][103] Ennek mutációja törékeny X-szindrómát, autizmust és más neurológiai betegségeket okozhat.[104] A törékeny X-szindrómát az FMR1 gén 13. exonjában lévő CGG ismétlődések számának 50-ről több mint 200-ra való növekedése okozza, elősegítve a DNS-metilációt és más epigenetikai heterokromatin-módosulásokat, melyek a gén transzkripcióját akadályozzák, alacsony FMRP-szinteket okozva.[105][106]
Terápiás módszerek
szerkesztésAz antiszenszmediált beavatkozások és a kis ligandumok gyakori stratégiák a G-kvadruplex-ismétlődésekhez kötődő neurológiai betegségekhez. Így ezek különösen hasznosak a funkciószerzéses neurológiai betegségek célzására, ahol a módosult gén terméke új funkcióval vagy a gén új expressziójával rendelkezik; ez szlelhető a C9orf72 (9. kromoszóma, 72. nyitott leolvasási keret) esetén is.[107]
Az antiszensz-terápia során szintetizált nukleinsavszálak kapcsolódnak közvetlenül és specifikusan egy gén mRNS-éhez, inaktiválva azt. Az antiszensz oligonukleotidok (ASO) gyakoriak a C9orf72 RNS célzására a GGGGCC ismétlődő szakaszban, csökkentve a C9orf72 sejtmodelljeiben a toxicitást.[108][109][110] Az ASO-kat korábban is használták funkciószerzéses neurológiai betegségekben az eredeti fenotípus helyreállítására, az egyetlen különbség az volt, hogy G-kvadruplex-ismétlődéses rész nélkül használták.[111][112][113][114]
A G-kvadruplex-csali egy másik ígéretes módszer ráksejtek ellen a G-kvadruplex szerkezetének kihasználására. Ez a G-kvadruplexhez hasonló szerkezetű és az endogén G-kvadruplexekkel a transzkripciós faktorhoz kötődésért versenyző szintetikus oligonukleotidokat használ. Ezek jellemzően egy stabil G-kvadruplex alkotására képes G-gazdag szekvenciából és egy rövid, tulajdonságaik optimalizálásához módosítható kötőrészből állnak.[115] A ráksejtekbe jutva a csali megzavarhatja és megkötheti a megfelelő transzkripciós faktorokat a génkifejeződés szabályozásához vezetve. A csalikat sikeresen használták az onkogén KRAS gátlására SCID-egerekben, csökkentve a tumornövekedést és növelve a medián túlélési időt.[116]
További gyakori módszer a kis ligandumok használata. Ezek használhatók neurológiai rendellenességeket okozó G-kvadruplexek célzására. Mintegy 1000 G-kvadruplex-ligandum van, melyekkel különböző módon kölcsönhatásba léphetnek aromás gyűrűik révén, lehetővé téve a kis ligandumoknak a síkalkatú terminális tetrádokhoz csatlakozást a G-kvadruplexekben. Ennek hátránya a nehezen elérhető specificitás a G-kvadruplexek változatos szekvenciái, elrendezése, stabilitása és nukleinsav-sztöchiometriája miatt. 2020 márciusáig nem ismert egy kis ligandum, mely egy adott G-kvadruplexre lenne specifikus.[117][118] Azonban egy kationos porfin, a TMPyP4 képes a C9orf72 GGGGCC-ismétlődéses szakaszához kapcsolódni, a G-kvadruplex-ismétlődéses szakasz széthajtását és a fehérjékkel való kölcsönhatásai és funkciója elvesztését okozva.[119] Kis, főleg ólomtartalmú ligandumok a GGGGCC részeket is célozhatják, csökkentve az ismétlődésasszociált nem ATG-transzlációs és az RNS-fókuszokat az ALS-sel élő betegek neuronjaiban. Így a kis ligandumok hatékonyak és hatásosak lehetnek a GGGGCC részekre, és e specificitás elérhető lehet kis ligandumok megkötésére is.
A fémkomplexek számos G4-DNS-kötésre alkalmassá és lehetséges gyógyszerré tevő jellemzővel rendelkeznek. Míg a fém főleg szerkezeti szereppel rendelkezik a legtöbb G4-kötőben, vannak esetek, mikor közvetlenül a G4-gyel lépnek kölcsönhatásba elektrosztatikusan vagy a nukleobázisokkal való közvetlen koordinációval.[120]
Jegyzetek
szerkesztés- ↑ J. A. Capra, K. Paeschke, M. Singh, V. A. Zakian (2010. július 1.). „G-quadruplex DNA sequences are evolutionarily conserved and associated with distinct genomic features in Saccharomyces cerevisiae”. PLOS Computational Biology 6 (7), e1000861. o. DOI:10.1371/journal.pcbi.1000861. PMID 20676380. PMC 2908698.
- ↑ Routh ED, Creacy SD, Beerbower PE, Akman SA, Vaughn JP, Smaldino PJ (2017. március 1.). „A G-quadruplex DNA-affinity Approach for Purification of Enzymaticacvly Active G4 Resolvase1”. Journal of Visualized Experiments 121 (121). DOI:10.3791/55496. PMID 28362374. PMC 5409278.
- ↑ a b E. Largy, J. Mergny, V. Gabelica.szerk.: S. Astrid, S. Helmut, K. O. Roland: Chapter 7. Role of Alkali Metal Ions in G-Quadruplex Nucleic Acid Structure and Stability, The Alkali Metal Ions: Their Role in Life, Metal Ions in Life Sciences. Springer, 203–258. o.. DOI: 10.1007/978-3-319-21756-7_7 (2016)
- ↑ a b W. I. Sundquist, A. Klug (1989. december 1.). „Telomeric DNA dimerizes by formation of guanine tetrads between hairpin loops”. Nature 342 (6251), 825–829. o. DOI:10.1038/342825a0. PMID 2601741.
- ↑ a b c Sen D, Gilbert W (1988. július 1.). „Formation of parallel four-stranded complexes by guanine-rich motifs in DNA and its implications for meiosis”. Nature 334 (6180), 364–366. o. DOI:10.1038/334364a0. PMID 3393228.
- ↑ a b c d Rawal P, Kummarasetti VB, Ravindran R, Kumar N, Halder K, Sharma R, Mukerji M, Das SK, Chowdhury S (2006. december 8.). „Genome-wide Prediction of G4 DNA as Regulatory Motifs: Role in Escherichia Coli Global Regulation”. Genome Research 16 (5), 644‐655. o. DOI:10.1101/gr.4508806. PMID 16651665. PMC 1457047.
- ↑ a b c d S. Borman (2007. május 28.). „Ascent of quadruplexes nucleic acid structures become promising drug targets”. Chemical and Engineering News 85 (22), 12–17. o. DOI:10.1021/cen-v085n009.p012a.
- ↑ Verma A, Halder K, Halder R, Yadav VK, Rawal P, Thakur RK, Mohd F, Sharma A, Chowdhury S (2008). „Genome-wide Computational and Expression Analyses Reveal G-quadruplex DNA Motifs as Conserved Cis-Regulatory Elements in Human and Related Species”. Journal of Medicinal Chemistry 51 (18), 5641‐5649. o. DOI:10.1021/jm800448a. PMID 18767830.
- ↑ H. Han, L. H. Hurley (2000. április 1.). „G-quadruplex DNA: a potential target for anti-cancer drug design”. Trends in Pharmacological Sciences 21 (4), 136–142. o. DOI:10.1016/s0165-6147(00)01457-7. PMID 10740289.
- ↑ M. L. Bochman, K. Paeschke, V. A. Zakian (2012. november 1.). „DNA secondary structures: stability and function of G-quadruplex structures”. Nature Reviews. Genetics 13 (11), 770–80. o. DOI:10.1038/nrg3296. PMID 23032257. PMC 3725559.
- ↑ Yadav VK, Abraham JK, Mani P, Kulshrestha R, Chowdhury S (2008. december 8.). „QuadBase: Genome-Wide Database of G4 DNA--occurrence and Conservation in Human, Chimpanzee, Mouse and Rat Promoters and 146 Microbes”. Nucleic Acids Research 36 (Database), D381‐D385. o. DOI:10.1093/nar/gkm781. PMID 17962308. PMC 2238983.
- ↑ P. Dhapola, S. Chowdhury (2016. július 1.). „QuadBase2: Web Server for Multiplexed Guanine Quadruplex Mining and Visualization”. Nucleic Acids Research 44 (W1), W277‐W283. o. DOI:10.1093/nar/gkw425. PMID 27185890. PMC 4987949.
- ↑ D. Rhodes, H. J. Lipps (2015. október 1.). „G-quadruplexes and their regulatory roles in biology”. Nucleic Acids Research 43 (18), 8627–37. o. DOI:10.1093/nar/gkv862. PMID 26350216. PMC 4605312.
- ↑ S. Borman (2009. november 1.). „Promoter quadruplexes folded DNA structures in gene-activation sites may be useful cancer drug targets”. Chemical and Engineering News 87 (44), 28–30. o. DOI:10.1021/cen-v087n044.p028.
- ↑ Gellert M, Lipsett MN, Davies DR (1962. december 1.). „Helix formation by guanylic acid”. Proceedings of the National Academy of Sciences of the United States of America 48 (12), 2013–2018. o. DOI:10.1073/pnas.48.12.2013. PMID 13947099. PMC 221115.
- ↑ Henderson E, Hardin CC, Walk SK, Tinoco I, Blackburn EH (1987. december 1.). „Telomeric DNA oligonucleotides form novel intramolecular structures containing guanine-guanine base pairs”. Cell 51 (6), 899–908. o. DOI:10.1016/0092-8674(87)90577-0. PMID 3690664.
- ↑ (2010. október 10.) „Small-molecule-mediated G-quadruplex isolation from human cells”. Nature Chemistry 2 (12), 1095–1098. o. DOI:10.1038/nchem.842. PMID 21107376. PMC 3119466.
- ↑ (2012. február 5.) „Small-molecule–induced DNA damage identifies alternative DNA structures in human genes”. Nature Chemical Biology 8 (3), 301–310. o. DOI:10.1038/nchembio.780. PMID 22306580. PMC 3433707.
- ↑ T. Simonsson (2001. április 1.). „G-quadruplex DNA structures--variations on a theme”. Biological Chemistry 382 (4), 621–8. o. DOI:10.1515/BC.2001.073. PMID 11405224.
- ↑ Burge S, Parkinson GN, Hazel P, Todd AK, Neidle S (2006). „Quadruplex DNA: sequence, topology and structure”. Nucleic Acids Research 34 (19), 5402–15. o. DOI:10.1093/nar/gkl655. PMID 17012276. PMC 1636468.
- ↑ Cao K, Ryvkin P, Johnson FB (2012. május 1.). „Computational detection and analysis of sequences with duplex-derived interstrand G-quadruplex forming potential”. Methods 57 (1), 3–10. o. DOI:10.1016/j.ymeth.2012.05.002. PMID 22652626. PMC 3701776.
- ↑ Kudlicki AS (2016). „G-Quadruplexes Involving Both Strands of Genomic DNA Are Highly Abundant and Colocalize with Functional Sites in the Human Genome”. PLOS ONE 11 (1), e0146174. o. DOI:10.1371/journal.pone.0146174. PMID 26727593. PMC 4699641.
- ↑ Murat P, Balasubramanian S (2014. április 1.). „Existence and consequences of G-quadruplex structures in DNA”. Current Opinion in Genetics & Development 25 (25), 22–29. o. DOI:10.1016/j.gde.2013.10.012. PMID 24584093.
- ↑ Miyoshi D, Karimata H, Sugimoto N (2006. június 1.). „Hydration regulates thermodynamics of G-quadruplex formation under molecular crowding conditions”. Journal of the American Chemical Society 128 (24), 7957–63. o. DOI:10.1021/ja061267m. PMID 16771510.
- ↑ Zheng KW, Chen Z, Hao YH, Tan Z (2010. január 1.). „Molecular crowding creates an essential environment for the formation of stable G-quadruplexes in long double-stranded DNA”. Nucleic Acids Research 38 (1), 327–38. o. DOI:10.1093/nar/gkp898. PMID 19858105. PMC 2800236.
- ↑ Endoh T, Rode AB, Takahashi S, Kataoka Y, Kuwahara M, Sugimoto N (2016. február 1.). „Real-Time Monitoring of G-Quadruplex Formation during Transcription”. Analytical Chemistry 88 (4), 1984–9. o. DOI:10.1021/acs.analchem.5b04396. PMID 26810457.
- ↑ Wang Q, Liu JQ, Chen Z, Zheng KW, Chen CY, Hao YH, Tan Z (2011. augusztus 1.). „G-quadruplex formation at the 3' end of telomere DNA inhibits its extension by telomerase, polymerase and unwinding by helicase”. Nucleic Acids Research 39 (14), 6229–37. o. DOI:10.1093/nar/gkr164. PMID 21441540. PMC 3152327.
- ↑ Schaffitzel C, Berger I, Postberg J, Hanes J, Lipps HJ, Plückthun A (2001. július 1.). „In vitro generated antibodies specific for telomeric guanine-quadruplex DNA react with Stylonychia lemnae macronuclei”. Proceedings of the National Academy of Sciences of the United States of America 98 (15), 8572–7. o. DOI:10.1073/pnas.141229498. PMID 11438689. PMC 37477.
- ↑ Paeschke K, Simonsson T, Postberg J, Rhodes D, Lipps HJ (2005. október 1.). „Telomere end-binding proteins control the formation of G-quadruplex DNA structures in vivo”. Nature Structural & Molecular Biology 12 (10), 847–54. o. DOI:10.1038/nsmb982. PMID 16142245.
- ↑ Kar A, Jones N, Arat NÖ, Fishel R, Griffith JD (2018. június 1.). „Long repeating (TTAGGG)n single-stranded DNA self-condenses into compact beaded filaments stabilized by G-quadruplex formation”. The Journal of Biological Chemistry 293 (24), 9473–9485. o. DOI:10.1074/jbc.RA118.002158. PMID 29674319. PMC 6005428.
- ↑ Richl T, Kuper J, Kisker C (2024. február 26.). „G-quadruplex-mediated genomic instability drives SNVs in cancer”. Nucleic Acids Res 52 (4), gkae098. o. DOI:10.1093/nar/gkae098. (Hozzáférés: 2024. február 26.)
- ↑ Volná A, Bartas M, Karlický V, Nezval J, Kundrátová K, Pečinka P, Špunda V, Červeň J (2021. július 1.). „G-Quadruplex in Gene Encoding Large Subunit of Plant RNA Polymerase II: A Billion-Year-Old Story”. International Journal of Molecular Sciences 22 (14), 7381. o. DOI:10.3390/ijms22147381. PMID 34299001. PMC 8306923.
- ↑ Simonsson T, Pecinka P, Kubista M (1998. március 1.). „DNA tetraplex formation in the control region of c-myc”. Nucleic Acids Research 26 (5), 1167–72. o. DOI:10.1093/nar/26.5.1167. PMID 9469822. PMC 147388.
- ↑ Siddiqui-Jain A, Grand CL, Bearss DJ, Hurley LH (2002. szeptember 1.). „Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription”. Proceedings of the National Academy of Sciences of the United States of America 99 (18), 11593–8. o. DOI:10.1073/pnas.182256799. PMID 12195017. PMC 129314.
- ↑ Huppert JL, Balasubramanian S (2006. december 14.). „G-quadruplexes in promoters throughout the human genome”. Nucleic Acids Research 35 (2), 406–13. o. DOI:10.1093/nar/gkl1057. PMID 17169996. PMC 1802602.
- ↑ Dai J, Dexheimer TS, Chen D, Carver M, Ambrus A, Jones RA, Yang D (2006. február 1.). „An intramolecular G-quadruplex structure with mixed parallel/antiparallel G-strands formed in the human BCL-2 promoter region in solution”. Journal of the American Chemical Society 128 (4), 1096–8. o. DOI:10.1021/ja055636a. PMID 16433524. PMC 2556172.
- ↑ a b Fernando H, Reszka AP, Huppert J, Ladame S, Rankin S, Venkitaraman AR, Neidle S, Balasubramanian S (2006. június 1.). „A conserved quadruplex motif located in a transcription activation site of the human c-kit oncogene”. Biochemistry 45 (25), 7854–60. o. DOI:10.1021/bi0601510. PMID 16784237. PMC 2195898.
- ↑ Huppert JL, Balasubramanian S (2005). „Prevalence of quadruplexes in the human genome”. Nucleic Acids Research 33 (9), 2908–16. o. DOI:10.1093/nar/gki609. PMID 15914667. PMC 1140081.
- ↑ Rawal P, Kummarasetti VB, Ravindran J, Kumar N, Halder K, Sharma R, Mukerji M, Das SK, Chowdhury S (2006. május 1.). „Genome-wide prediction of G4 DNA as regulatory motifs: role in Escherichia coli global regulation”. Genome Research 16 (5), 644–55. o. DOI:10.1101/gr.4508806. PMID 16651665. PMC 1457047.
- ↑ Kamath-Loeb A, Loeb LA, Fry M (2012). „The Werner syndrome protein is distinguished from the Bloom syndrome protein by its capacity to tightly bind diverse DNA structures”. PLOS ONE 7 (1), e30189. o. DOI:10.1371/journal.pone.0030189. PMID 22272300. PMC 3260238.
- ↑ Vaughn JP, Creacy SD, Routh ED, Joyner-Butt C, Jenkins GS, Pauli S, Nagamine Y, Akman SA (2005. november 1.). „The DEXH protein product of the DHX36 gene is the major source of tetramolecular quadruplex G4-DNA resolving activity in HeLa cell lysates”. The Journal of Biological Chemistry 280 (46), 38117–20. o. DOI:10.1074/jbc.C500348200. PMID 16150737.
- ↑ Chen MC, Ferré-D'Amaré AR (2017. augusztus 15.). „Structural Basis of DEAH/RHA Helicase Activity”. Crystals 7 (8), 253. o. DOI:10.3390/cryst7080253.
- ↑ a b Thakur RK, Kumar P, Halder K, Verma A, Kar A, Parent JL, Basundra R, Kumar A, Chowdhury S (2009. január 1.). „Metastases Suppressor NM23-H2 Interaction With G-quadruplex DNA Within c-MYC Promoter Nuclease Hypersensitive Element Induces c-MYC Expression”. Nucleic Acids Research 37 (1), 172‐183. o. DOI:10.1093/nar/gkn919. PMID 19033359. PMC 2615625.
- ↑ Borman S (2009. november 1.). „Promoter Quadruplexes Folded DNA structures in gene-activation sites may be useful cancer drug targets”. Chemical and Engineering News 87 (44), 28–30. o. DOI:10.1021/cen-v087n044.p028.
- ↑ a b c Saha D, Singh A, Hussain T, Srivastava V, Sengupta S, Kar A, Dhapola P, Ummanni R, Chowdhury S (2017. július 1.). „Epigenetic Suppression of Human Telomerase ( hTERT) Is Mediated by the Metastasis Suppressor NME2 in a G-quadruplex-dependent Fashion”. The Journal of Biological Chemistry 292 (37), 15205‐15215. o. DOI:10.1074/jbc.M117.792077. PMID 28717007. PMC 5602382.
- ↑ Mukherjee AK, Sharma S, Bagri S, Kutum R, Kumar P, Hussain A, Singh P, Saha D, Kar A, Dash D, Chowdhury S (2019. november 1.). „Telomere Repeat-Binding Factor 2 Binds Extensively to Extra-Telomeric G-quadruplexes and Regulates the Epigenetic Status of Several Gene Promoters”. The Journal of Biological Chemistry 294 (47), 17709–17722. o. DOI:10.1074/jbc.RA119.008687. PMID 31575660. PMC 6879327.
- ↑ Maizels N, Gray LT (2013. április 1.). „The G4 genome”. PLOS Genetics 9 (4), e1003468. o. DOI:10.1371/journal.pgen.1003468. PMID 23637633. PMC 3630100.
- ↑ Biffi G, Tannahill D, McCafferty J, Balasubramanian S (2013. március 1.). „Quantitative visualization of DNA G-quadruplex structures in human cells”. Nature Chemistry 5 (3), 182–6. o. DOI:10.1038/nchem.1548. PMID 23422559. PMC 3622242.
- ↑ Chen MC, Tippana R, Demeshkina NA, Murat P, Balasubramanian S, Myong S, Ferré-D'Amaré AR (2018. június 1.). „Structural basis of G-quadruplex unfolding by the DEAH/RHA helicase DHX36” (angol nyelven). Nature 558 (7710), 465–469. o. DOI:10.1038/s41586-018-0209-9. PMID 29899445. PMC 6261253.
- ↑ Rice C, Skordalakes E (2016). „Structure and function of the telomeric CST complex”. Computational and Structural Biotechnology Journal 14, 161–7. o. DOI:10.1016/j.csbj.2016.04.002. PMID 27239262. PMC 4872678.
- ↑ Hänsel-Hertsch R, Beraldi D, Lensing SV, Marsico G, Zyner K, Parry A, Di Antonio M, Pike J, Kimura H, Narita M, Tannahill D, Balasubramanian S (2016. október 1.). „G-quadruplex structures mark human regulatory chromatin”. Nature Genetics 48 (10), 1267–1272. o. DOI:10.1038/ng.3662. PMID 27618450.
- ↑ a b Poetsch AR. AP-Seq: A Method to Measure Apurinic Sites and Small Base Adducts Genome-Wide, The Nucleus, Methods in Molecular Biology, 95–108. o.. DOI: 10.1007/978-1-0716-0763-3_8 (2020. december 8.). ISBN 978-1-0716-0762-6
- ↑ a b c d e f g Roychoudhury S, Pramanik S, Harris HL, Tarpley M, Sarkar A, Spagnol G, Sorgen PL, Chowdhury D, Band V, Klinkebiel D, Bhakat KK (2020. május 1.). „Endogenous oxidized DNA bases and APE1 regulate the formation of G-quadruplex structures in the genome”. Proceedings of the National Academy of Sciences of the United States of America 117 (21), 11409–11420. o. DOI:10.1073/pnas.1912355117. PMID 32404420. PMC 7260947.
- ↑ a b c d Canugovi C, Shamanna RA, Croteau DL, Bohr VA (2014. június 1.). „Base excision DNA repair levels in mitochondrial lysates of Alzheimer's disease”. Neurobiology of Aging 35 (6), 1293–1300. o. DOI:10.1016/j.neurobiolaging.2014.01.004. PMID 24485507. PMC 5576885.
- ↑ a b Sun D, Hurley LH (2009. május 1.). „The importance of negative superhelicity in inducing the formation of G-quadruplex and i-motif structures in the c-Myc promoter: implications for drug targeting and control of gene expression”. Journal of Medicinal Chemistry 52 (9), 2863–2874. o. DOI:10.1021/jm900055s. PMID 19385599. PMC 2757002.
- ↑ a b Hill JW, Hazra TK, Izumi T, Mitra S (2001. január 1.). „Stimulation of human 8-oxoguanine-DNA glycosylase by AP-endonuclease: potential coordination of the initial steps in base excision repair”. Nucleic Acids Research 29 (2), 430–438. o. DOI:10.1093/nar/29.2.430. PMID 11139613. PMC 29662.
- ↑ C. J. Burrows, J. G. Muller (1998. május 1.). „Oxidative Nucleobase Modifications Leading to Strand Scission”. Chemical Reviews 98 (3), 1109–1152. o. DOI:10.1021/cr960421s. PMID 11848927.
- ↑ a b c d A. R. Poetsch (2020. január 7.). „The genomics of oxidative DNA damage, repair, and resulting mutagenesis”. Computational and Structural Biotechnology Journal 18, 207–219. o. DOI:10.1016/j.csbj.2019.12.013. PMID 31993111. PMC 6974700.
- ↑ a b Fleming AM, Burrows CJ (2017. október 1.). „8-Oxo-7,8-dihydro-2'-deoxyguanosine and abasic site tandem lesions are oxidation prone yielding hydantoin products that strongly destabilize duplex DNA”. Organic & Biomolecular Chemistry 15 (39), 8341–8353. o. DOI:10.1039/C7OB02096A. PMID 28936535. PMC 5636683.
- ↑ a b Kitsera N, Rodriguez-Alvarez M, Emmert S, Carell T, Khobta A (2019. szeptember 1.). „Nucleotide excision repair of abasic DNA lesions”. Nucleic Acids Research 47 (16), 8537–8547. o. DOI:10.1093/nar/gkz558. PMID 31226203. PMC 6895268.
- ↑ Roychoudhury S, Nath S, Song H, Hegde ML, Bellot LJ, Mantha AK, Sengupta S, Ray S, Natarajan A, Bhakat KK (2017. március 1.). „Human Apurinic/Apyrimidinic Endonuclease (APE1) Is Acetylated at DNA Damage Sites in Chromatin, and Acetylation Modulates Its DNA Repair Activity”. Molecular and Cellular Biology 37 (6). DOI:10.1128/mcb.00401-16. PMID 27994014. PMC 5335514.
- ↑ Chattopadhyay R, Das S, Maiti AK, Boldogh I, Xie J, Hazra TK, Kohno K, Mitra S, Bhakat KK (2008. december 1.). „Regulatory role of human AP-endonuclease (APE1/Ref-1) in YB-1-mediated activation of the multidrug resistance gene MDR1”. Molecular and Cellular Biology 28 (23), 7066–7080. o. DOI:10.1128/mcb.00244-08. PMID 18809583. PMC 2593380.
- ↑ Bhakat KK, Izumi T, Yang SH, Hazra TK, Mitra S (2003. december 1.). „Role of acetylated human AP-endonuclease (APE1/Ref-1) in regulation of the parathyroid hormone gene”. The EMBO Journal 22 (23), 6299–6309. o. DOI:10.1093/emboj/cdg595. PMID 14633989. PMC 291836.
- ↑ Yamamori T, DeRicco J, Naqvi A, Hoffman TA, Mattagajasingh I, Kasuno K, Jung SB, Kim CS, Irani K (2010. január 1.). „SIRT1 deacetylates APE1 and regulates cellular base excision repair”. Nucleic Acids Research 38 (3), 832–845. o. DOI:10.1093/nar/gkp1039. PMID 19934257. PMC 2817463.
- ↑ Neidle S (2016. július 1.). „Quadruplex Nucleic Acids as Novel Therapeutic Targets” (angol nyelven). Journal of Medicinal Chemistry 59 (13), 5987–6011. o. DOI:10.1021/acs.jmedchem.5b01835. PMID 26840940.
- ↑ Y. Chen, D. Yang. Sequence, stability, and structure of G-quadruplexes and their interactions with drugs, 17.5.1–17.5.17. o. (2012. szeptember 1.). ISBN 978-0471142706
- ↑ T. A. Brooks, S. Kendrick, L. Hurley (2010. szeptember 1.). „Making sense of G-quadruplex and i-motif functions in oncogene promoters”. The FEBS Journal 277 (17), 3459–69. o. DOI:10.1111/j.1742-4658.2010.07759.x. PMID 20670278. PMC 2971675.
- ↑ a b Ou TM, Lin J, Lu YJ, Hou JQ, Tan JH, Chen SH, Li Z, Li YP, Li D, Gu LQ, Huang ZS (2011. augusztus 1.). „Inhibition of cell proliferation by quindoline derivative (SYUIQ-05) through its preferential interaction with c-myc promoter G-quadruplex”. Journal of Medicinal Chemistry 54 (16), 5671–9. o. DOI:10.1021/jm200062u. PMID 21774525.
- ↑ Sharma S, Mukherjee AK, Roy SS, Bagri S, Lier S, Verma M, Sengupta A, Kumar M, Nesse G, Pandey DP, Chowdhury S (2020. január 1.). „Human Telomerase Expression is under Direct Transcriptional Control of the Telomere-binding-factor TRF2”. bioRxiv. DOI:10.1101/2020.01.15.907626.
- ↑ Sun D, Guo K, Rusche JJ, Hurley LH (2005. október 12.). „Facilitation of a structural transition in the polypurine/polypyrimidine tract within the proximal promoter region of the human VEGF gene by the presence of potassium and G-quadruplex-interactive agents”. Nucleic Acids Research 33 (18), 6070–80. o. DOI:10.1093/nar/gki917. PMID 16239639. PMC 1266068.
- ↑ Hussain T, Saha D, Purohit G, Mukherjee AK, Sharma S, Sengupta S, Dhapola P, Maji B, Vedagopuram S, Horikoshi NT, Horikoshi N, Pandita RK, Bhattacharya S, Bajaj A, Riou JF, Pandita TK, Chowdhury S (2017. szeptember 1.). „Transcription Regulation of CDKN1A (p21/CIP1/WAF1) by TRF2 Is Epigenetically Controlled Through the REST Repressor Complex”. Scientific Reports 7 (1), 11541. o. DOI:10.1038/s41598-017-11177-1. PMID 28912501. PMC 5599563.
- ↑ De Armond R, Wood S, Sun D, Hurley LH, Ebbinghaus SW (2005. december 1.). „Evidence for the presence of a guanine quadruplex forming region within a polypurine tract of the hypoxia inducible factor 1alpha promoter”. Biochemistry 44 (49), 16341–50. o. DOI:10.1021/bi051618u. PMID 16331995.
- ↑ Guo K, Pourpak A, Beetz-Rogers K, Gokhale V, Sun D, Hurley LH (2007. augusztus 1.). „Formation of pseudosymmetrical G-quadruplex and i-motif structures in the proximal promoter region of the RET oncogene”. Journal of the American Chemical Society 129 (33), 10220–8. o. DOI:10.1021/ja072185g. PMID 17672459. PMC 2566970.
- ↑ Qin Y, Rezler EM, Gokhale V, Sun D, Hurley LH (2007. november 26.). „Characterization of the G-quadruplexes in the duplex nuclease hypersensitive element of the PDGF-A promoter and modulation of PDGF-A promoter activity by TMPyP4”. Nucleic Acids Research 35 (22), 7698–713. o. DOI:10.1093/nar/gkm538. PMID 17984069. PMC 2190695.
- ↑ Chilakamarthi U, Koteshwar D, Jinka S, Vamsi Krishna N, Sridharan K, Nagesh N, Giribabu L (2018. november 1.). „Novel Amphiphilic G-Quadruplex Binding Synthetic Derivative of TMPyP4 and Its Effect on Cancer Cell Proliferation and Apoptosis Induction”. Biochemistry 57 (46), 6514–6527. o. DOI:10.1021/acs.biochem.8b00843. PMID 30369235.
- ↑ Ohnmacht SA, Marchetti C, Gunaratnam M, Besser RJ, Haider SM, Di Vita G, Lowe HL, Mellinas-Gomez M, Diocou S, Robson M, Šponer J, Islam B, Pedley RB, Hartley JA, Neidle S (2015. június 1.). „A G-quadruplex-binding compound showing anti-tumour activity in an in vivo model for pancreatic cancer”. Scientific Reports 5, 11385. o. DOI:10.1038/srep11385. PMID 26077929. PMC 4468576.
- ↑ Siddiqui-Jain A, Grand CL, Bearss DJ, Hurley LH (2002. szeptember 1.). „Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription”. Proceedings of the National Academy of Sciences of the United States of America 99 (18), 11593–8. o. DOI:10.1073/pnas.182256799. PMID 12195017. PMC 129314.
- ↑ Collie GW, Promontorio R, Hampel SM, Micco M, Neidle S, Parkinson GN (2012. február 1.). „Structural basis for telomeric G-quadruplex targeting by naphthalene diimide ligands” (angol nyelven). Journal of the American Chemical Society 134 (5), 2723–31. o. DOI:10.1021/ja2102423. PMID 22280460.
- ↑ Todd AK, Johnston M, Neidle S (2005). „Highly prevalent putative quadruplex sequence motifs in human DNA”. Nucleic Acids Research 33 (9), 2901–7. o. DOI:10.1093/nar/gki553. PMID 15914666. PMC 1140077.
- ↑ Frank-Kamenetskii MD, Mirkin SM (1995). „Triplex DNA structures”. Annual Review of Biochemistry 64 (9), 65–95. o. DOI:10.1146/annurev.bi.64.070195.000433. PMID 7574496.
- ↑ Guo K, Gokhale V, Hurley LH, Sun D (2008. augusztus 1.). „Intramolecularly folded G-quadruplex and i-motif structures in the proximal promoter of the vascular endothelial growth factor gene”. Nucleic Acids Research 36 (14), 4598–608. o. DOI:10.1093/nar/gkn380. PMID 18614607. PMC 2504309.
- ↑ Mirkin SM, Lyamichev VI, Drushlyak KN, Dobrynin VN, Filippov SA, Frank-Kamenetskii MD (1987). „DNA H form requires a homopurine-homopyrimidine mirror repeat”. Nature 330 (6147), 495–7. o. DOI:10.1038/330495a0. PMID 2825028.
- ↑ S. S. Smith (2010). „Evolutionary expansion of structurally complex DNA sequences”. Cancer Genomics & Proteomics 7 (4), 207–15. o. PMID 20656986.
- ↑ (2019. szeptember 15.) „G4Hunter web application: a web server for G-quadruplex prediction” (angol nyelven). Bioinformatics 35 (18), 3493–3495. o. DOI:10.1093/bioinformatics/btz087. ISSN 1367-4803. PMID 30721922. PMC 6748775.
- ↑ (2018) „G4RNA screener web server: User focused interface for RNA G-quadruplex prediction” (angol nyelven). Biochimie 151, 115–118. o. DOI:10.1016/j.biochi.2018.06.002. PMID 29885355.
- ↑ C. K. Kwok, C. J. Merrick (2017. október 1.). „G-Quadruplexes: Prediction, Characterization, and Biological Application”. Trends in Biotechnology 35 (10), 997–1013. o. DOI:10.1016/j.tibtech.2017.06.012. PMID 28755976.
- ↑ H. Han, L. H. Hurley, M. Salazar (1999. január 1.). „A DNA polymerase stop assay for G-quadruplex-interactive compounds”. Nucleic Acids Research 27 (2), 537–542. o. DOI:10.1093/nar/27.2.537. PMID 9862977. PMC 148212.
- ↑ Sun D, Hurley LH. Biochemical Techniques for the Characterization of G-Quadruplex Structures: EMSA, DMS Footprinting, and DNA Polymerase Stop Assay, G-Quadruplex DNA, Methods in Molecular Biology. Humana Press, 65–79. o.. DOI: 10.1007/978-1-59745-363-9_5 (2009. október 23.). ISBN 9781588299505
- ↑ S- Paramasovan, I. Rujan, P. H. Bolton (2007. december 1.). „Circular dichroism of quadruplex DNAs: applications to structure, cation effects and ligand binding”. Methods 43 (4), 324–331. o. DOI:10.1016/j.ymeth.2007.02.009. PMID 17967702.
- ↑ Mergny JL, Phan AT, Lacroix L (1998. szeptember 1.). „Following G-quartet formation by UV-spectroscopy”. FEBS Letters 435 (1), 74–78. o. DOI:10.1016/s0014-5793(98)01043-6. PMID 9755862.
- ↑ An N, Fleming AM, Middleton EG, Burrows CJ (2014. október 1.). „Single-molecule investigation of G-quadruplex folds of the human telomere sequence in a protein nanocavity”. Proceedings of the National Academy of Sciences of the United States of America 111 (40), 14325–14331. o. DOI:10.1073/pnas.1415944111. PMID 25225404. PMC 4209999.
- ↑ Bošković F, Zhu J, Chen K, Keyser UF (2019. november 1.). „Monitoring G-Quadruplex Formation with DNA Carriers and Solid-State Nanopores”. Nano Letters 19 (11), 7996–8001. o. DOI:10.1021/acs.nanolett.9b03184. PMID 31577148.
- ↑ Simone R, Fratta P, Neidle S, Parkinson GN, Isaacs AM (2015. június 1.). „G-quadruplexes: Emerging roles in neurodegenerative diseases and the non-coding transcriptome”. FEBS Letters 589 (14), 1653–68. o. DOI:10.1016/j.febslet.2015.05.003. PMID 25979174.
- ↑ C9orf72 chromosome 9 open reading frame 72 [Homo sapiens] - Gene]. National Center for Biotechnology Information. U.S. National Library of Medicine
- ↑ Ratnavalli E, Brayne C, Dawson K, Hodges JR (2002. június 1.). „The prevalence of frontotemporal dementia”. Neurology 58 (11), 1615–21. o. DOI:10.1212/WNL.58.11.1615. PMID 12058088.
- ↑ Rutherford NJ, Heckman MG, Dejesus-Hernandez M, Baker MC, Soto-Ortolaza AI, Rayaprolu S, Stewart H, Finger E, Volkening K, Seeley WW, Hatanpaa KJ, Lomen-Hoerth C, Kertesz A, Bigio EH, Lippa C, Knopman DS, Kretzschmar HA, Neumann M, Caselli RJ, White CL, Mackenzie IR, Petersen RC, Strong MJ, Miller BL, Boeve BF, Uitti RJ, Boylan KB, Wszolek ZK, Graff-Radford NR, Dickson DW, Ross OA, Rademakers R (2012. december 1.). „Length of normal alleles of C9ORF72 GGGGCC repeat do not influence disease phenotype”. Neurobiology of Aging 33 (12), 2950.e5–7. o. DOI:10.1016/j.neurobiolaging.2012.07.005. PMID 22840558. PMC 3617405.
- ↑ Beck J, Poulter M, Hensman D, Rohrer JD, Mahoney CJ, Adamson G, Campbell T, Uphill J, Borg A, Fratta P, Orrell RW, Malaspina A, Rowe J, Brown J, Hodges J, Sidle K, Polke JM, Houlden H, Schott JM, Fox NC, Rossor MN, Tabrizi SJ, Isaacs AM, Hardy J, Warren JD, Collinge J, Mead S (2013. március 1.). „Large C9orf72 hexanucleotide repeat expansions are seen in multiple neurodegenerative syndromes and are more frequent than expected in the UK population”. American Journal of Human Genetics 92 (3), 345–353. o. DOI:10.1016/j.ajhg.2013.01.011. PMID 23434116. PMC 3591848.
- ↑ Fratta P, Mizielinska S, Nicoll AJ, Zloh M, Fisher EM, Parkinson G, Isaacs AM (2012. december 1.). „C9orf72 hexanucleotide repeat associated with amyotrophic lateral sclerosis and frontotemporal dementia forms RNA G-quadruplexes”. Scientific Reports 2, 1016. o. DOI:10.1038/srep01016. PMID 23264878. PMC 3527825.
- ↑ Reddy K, Zamiri B, Stanley SY, Macgregor RB, Pearson CE (2013. április 1.). „The disease-associated r(GGGGCC)n repeat from the C9orf72 gene forms tract length-dependent uni- and multimolecular RNA G-quadruplex structures”. The Journal of Biological Chemistry 288 (14), 9860–9866. o. DOI:10.1074/jbc.C113.452532. PMID 23423380. PMC 3617286.
- ↑ Haeusler AR, Donnelly CJ, Periz G, Simko EA, Shaw PG, Kim MS, Maragakis NJ, Troncoso JC, Pandey A, Sattler R, Rothstein JD, Wang J (2014. március 1.). „C9orf72 nucleotide repeat structures initiate molecular cascades of disease”. Nature 507 (7491), 195–200. o. DOI:10.1038/nature13124. PMID 24598541. PMC 4046618.
- ↑ Darnell JC, Jensen KB, Jin P, Brown V, Warren ST, Darnell RB (2001. november 1.). „Fragile X mental retardation protein targets G quartet mRNAs important for neuronal function”. Cell 107 (4), 489–499. o. DOI:10.1016/S0092-8674(01)00566-9. PMID 11719189.
- ↑ Ceman S, O'Donnell WT, Reed M, Patton S, Pohl J, Warren ST (2003. december 1.). „Phosphorylation influences the translation state of FMRP-associated polyribosomes”. Human Molecular Genetics 12 (24), 3295–3305. o. DOI:10.1093/hmg/ddg350. PMID 14570712.
- ↑ Fähling M, Mrowka R, Steege A, Kirschner KM, Benko E, Förstera B, Persson PB, Thiele BJ, Meier JC, Scholz H (2009. február 1.). „Translational regulation of the human achaete-scute homologue-1 by fragile X mental retardation protein”. The Journal of Biological Chemistry 284 (7), 4255–4266. o. DOI:10.1074/jbc.M807354200. PMID 19097999.
- ↑ Fragile X Mental Retardation. Gene Cards
- ↑ Pieretti M, Zhang FP, Fu YH, Warren ST, Oostra BA, Caskey CT, Nelson DL (1991. augusztus 1.). „Absence of expression of the FMR-1 gene in fragile X syndrome”. Cell 66 (4), 817–822. o. DOI:10.1016/0092-8674(91)90125-I. PMID 1878973.
- ↑ Sutcliffe JS, Nelson DL, Zhang F, Pieretti M, Caskey CT, Saxe D, Warren ST (1992. szeptember 1.). „DNA methylation represses FMR-1 transcription in fragile X syndrome”. Human Molecular Genetics 1 (6), 397–400. o. DOI:10.1093/hmg/1.6.397. PMID 1301913.
- ↑ S. Mizielinska, A. M. Isaacs (2014. október 1.). „C9orf72 amyotrophic lateral sclerosis and frontotemporal dementia: gain or loss of function?”. Current Opinion in Neurology 27 (5), 515–523. o. DOI:10.1097/WCO.0000000000000130. PMID 25188012. PMC 4165481.
- ↑ Donnelly CJ, Zhang PW, Pham JT, Haeusler AR, Heusler AR, Mistry NA, Vidensky S, Daley EL, Poth EM, Hoover B, Fines DM, Maragakis N, Tienari PJ, Petrucelli L, Traynor BJ, Wang J, Rigo F, Bennett CF, Blackshaw S, Sattler R, Rothstein JD (2013. október 1.). „RNA toxicity from the ALS/FTD C9ORF72 expansion is mitigated by antisense intervention”. Neuron 80 (2), 415–28. o. DOI:10.1016/j.neuron.2013.10.015. PMID 24139042. PMC 4098943.
- ↑ Lagier-Tourenne C, Baughn M, Rigo F, Sun S, Liu P, Li HR, Jiang J, Watt AT, Chun S, Katz M, Qiu J, Sun Y, Ling SC, Zhu Q, Polymenidou M, Drenner K, Artates JW, McAlonis-Downes M, Markmiller S, Hutt KR, Pizzo DP, Cady J, Harms MB, Baloh RH, Vandenberg SR, Yeo GW, Fu XD, Bennett CF, Cleveland DW, Ravits J (2013. november 1.). „Targeted degradation of sense and antisense C9orf72 RNA foci as therapy for ALS and frontotemporal degeneration”. Proceedings of the National Academy of Sciences of the United States of America 110 (47), E4530–9. o. DOI:10.1073/pnas.1318835110. PMID 24170860. PMC 3839752.
- ↑ Sareen D, O'Rourke JG, Meera P, Muhammad AK, Grant S, Simpkinson M, Bell S, Carmona S, Ornelas L, Sahabian A, Gendron T, Petrucelli L, Baughn M, Ravits J, Harms MB, Rigo F, Bennett CF, Otis TS, Svendsen CN, Baloh RH (2013. október 1.). „Targeting RNA foci in iPSC-derived motor neurons from ALS patients with a C9ORF72 repeat expansion”. Science Translational Medicine 5 (208), 208ra149. o. DOI:10.1126/scitranslmed.3007529. PMID 24154603. PMC 4090945.
- ↑ Wheeler TM, Leger AJ, Pandey SK, MacLeod AR, Nakamori M, Cheng SH, Wentworth BM, Bennett CF, Thornton CA (2012. augusztus 1.). „Targeting nuclear RNA for in vivo correction of myotonic dystrophy”. Nature 488 (7409), 111–5. o. DOI:10.1038/nature11362. PMID 22859208. PMC 4221572.
- ↑ Lee JE, Bennett CF, Cooper TA (2012. március 1.). „RNase H-mediated degradation of toxic RNA in myotonic dystrophy type 1”. Proceedings of the National Academy of Sciences of the United States of America 109 (11), 4221–6. o. DOI:10.1073/pnas.1117019109. PMID 22371589. PMC 3306674.
- ↑ Carroll JB, Warby SC, Southwell AL, Doty CN, Greenlee S, Skotte N, Hung G, Bennett CF, Freier SM, Hayden MR (2011. december 1.). „Potent and selective antisense oligonucleotides targeting single-nucleotide polymorphisms in the Huntington disease gene / allele-specific silencing of mutant huntingtin”. Molecular Therapy 19 (12), 2178–85. o. DOI:10.1038/mt.2011.201. PMID 21971427. PMC 3242664.
- ↑ Gagnon KT, Pendergraff HM, Deleavey GF, Swayze EE, Potier P, Randolph J, Roesch EB, Chattopadhyaya J, Damha MJ, Bennett CF, Montaillier C, Lemaitre M, Corey DR (2010. november 1.). „Allele-selective inhibition of mutant huntingtin expression with antisense oligonucleotides targeting the expanded CAG repeat”. Biochemistry 49 (47), 10166–78. o. DOI:10.1021/bi101208k. PMID 21028906. PMC 2991413.
- ↑ Cogoi S, Paramasivam M, Filichev V, Géci I, Pedersen EB, Xodo LE (2009. január 1.). „Identification of a new G-quadruplex motif in the KRAS promoter and design of pyrene-modified G4-decoys with antiproliferative activity in pancreatic cancer cells”. Journal of Medicinal Chemistry 52 (2), 564–568. o. DOI:10.1021/jm800874t. PMID 19099510.
- ↑ Cogoi S, Zorzet S, Rapozzi V, Géci I, Pedersen EB, Xodo LE (2013. április 1.). „MAZ-binding G4-decoy with locked nucleic acid and twisted intercalating nucleic acid modifications suppresses KRAS in pancreatic cancer cells and delays tumor growth in mice”. Nucleic Acids Research 41 (7), 4049–4064. o. DOI:10.1093/nar/gkt127. PMID 23471001. PMC 3627599.
- ↑ Campbell NH, Patel M, Tofa AB, Ghosh R, Parkinson GN, Neidle S (2009. március 1.). „Selectivity in ligand recognition of G-quadruplex loops”. Biochemistry 48 (8), 1675–80. o. DOI:10.1021/bi802233v. PMID 19173611.
- ↑ Ohnmacht SA, Neidle S (2014. június 1.). „Small-molecule quadruplex-targeted drug discovery”. Bioorganic & Medicinal Chemistry Letters 24 (12), 2602–12. o. DOI:10.1016/j.bmcl.2014.04.029. PMID 24814531.
- ↑ Zamiri B, Reddy K, Macgregor RB, Pearson CE (2014. február 1.). „TMPyP4 porphyrin distorts RNA G-quadruplex structures of the disease-associated r(GGGGCC)n repeat of the C9orf72 gene and blocks interaction of RNA-binding proteins”. The Journal of Biological Chemistry 289 (8), 4653–9. o. DOI:10.1074/jbc.C113.502336. PMID 24371143. PMC 3931028.
- ↑ R. Vilar.szerk.: A. Sigel, H. Sigel, E. Freisinger, R. K. Sigel: Chapter 12. Nucleic Acid Quadruplexes and Metallo-Drugs, Metallo-Drugs: Development and Action of Anticancer Agents, 325–349. o.. DOI: 10.1515/9783110470734-018 (2018). ISBN 9783110470734
Fordítás
szerkesztésEz a szócikk részben vagy egészben a G-quadruplex című angol Wikipédia-szócikk ezen változatának fordításán alapul. Az eredeti cikk szerkesztőit annak laptörténete sorolja fel. Ez a jelzés csupán a megfogalmazás eredetét és a szerzői jogokat jelzi, nem szolgál a cikkben szereplő információk forrásmegjelöléseként.
Források
szerkesztés- Ren J, Wang J, Han L, Wang E, Wang J (2011. október 1.). „Kinetically grafting G-quadruplexes onto DNA nanostructures for structure and function encoding via a DNA machine”. Chemical Communications 47 (38), 10563–5. o. DOI:10.1039/c1cc13973h. PMID 21858307.
- Johnson JE, Smith JS, Kozak ML, Johnson FB (2008. augusztus 1.). „In vivo veritas: using yeast to probe the biological functions of G-quadruplexes”. Biochimie 90 (8), 1250–63. o. DOI:10.1016/j.biochi.2008.02.013. PMID 18331848. PMC 2585026.
- J. L. Huppert, S. Balasubramanian (2005). „Prevalence of quadruplexes in the human genome”. Nucleic Acids Research 33 (9), 2908–16. o. DOI:10.1093/nar/gki609. PMID 15914667. PMC 1140081.
- A. K. Todd, M. Johnston, S. Neidle (2005). „Highly prevalent putative quadruplex sequence motifs in human DNA”. Nucleic Acids Research 33 (9), 2901–7. o. DOI:10.1093/nar/gki553. PMID 15914666. PMC 1140077.
- Burge S, Parkinson GN, Hazel P, Todd AK, Neidle S (2006). „Quadruplex DNA: sequence, topology and structure”. Nucleic Acids Research 34 (19), 5402–15. o. DOI:10.1093/nar/gkl655. PMID 17012276. PMC 1636468.
- Siddiqui-Jain A, Grand CL, Bearss DJ, Hurley LH (2002. szeptember 1.). „Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription”. Proceedings of the National Academy of Sciences of the United States of America 99 (18), 11593–8. o. DOI:10.1073/pnas.182256799. PMID 12195017. PMC 129314.
- Rawal P, Kummarasetti VB, Ravindran J, Kumar N, Halder K, Sharma R, Mukerji M, Das SK, Chowdhury S (2006. május 1.). „Genome-wide prediction of G4 DNA as regulatory motifs: role in Escherichia coli global regulation”. Genome Research 16 (5), 644–55. o. DOI:10.1101/gr.4508806. PMID 16651665. PMC 1457047.
- Hou X, Guo W, Xia F, Nie FQ, Dong H, Tian Y, Wen L, Wang L, Cao L, Yang Y, Xue J, Song Y, Wang Y, Liu D, Jiang L (2009. június 1.). „A biomimetic potassium responsive nanochannel: G-quadruplex DNA conformational switching in a synthetic nanopore”. Journal of the American Chemical Society 131 (22), 7800–5. o. DOI:10.1021/ja901574c. PMID 19435350.
- szerk.: Neidle & Balasubramanian: Quadruplex Nucleic Acids [archivált változat]. Royal Society of Chemistry (2006). ISBN 978-0-85404-374-3. Hozzáférés ideje: 2023. október 26. [archiválás ideje: 2007. szeptember 30.]
- Rowland GB, Barnett K, Dupont JI, Akurathi G, Le VH, Lewis EA (2013. december 1.). „The effect of pyridyl substituents on the thermodynamics of porphyrin binding to G-quadruplex DNA”. Bioorganic & Medicinal Chemistry 21 (23), 7515–22. o. DOI:10.1016/j.bmc.2013.09.036. PMID 24148836.
További információk
szerkesztés- Nanopore and Aptamer Biosensor group{NAB group}
- G-Quadruplex World – a G-kvadruplexekről szóló tanulmányok és más információk megbeszéléséről szóló lap
- Greglist – G-kvadruplex-szabályozott gének adatbázisa
- Database on Quadruplex information: QuadBase from IGIB
- GRSDB – RNS-feldolgozó helyekhez közeli G-kvadruplexek adatbázisa.
- GRS_UTRdb Archiválva 2011. július 20-i dátummal a Wayback Machine-ben. – UTR-beli G-kvadruplexek adatbázisa.
- G-quadruplex Resource Site
- non-B Motif Search Tool at non-B DB – G-kvadruplex- és más nem B-DNS-alkotó szekvenciák előrejelzése DNS-szekvenciákból
- QGRS Mapper: a web-based application for predicting G-quadruplexes in nucleotide sequences and NCBI genes.
- Quadfinder: Tool for Prediction and Analysis of G Quadruplex Motifs in DNA/RNA Sequences from Maiti's group, IGIB, Delhi, India[halott link]
- G4Hunter
- pqsfinder
- pqsfinder: onilne kereső a legfrissebb R/Bioconductor-csomaggal
| Az itt található információk kizárólag tájékoztató jellegűek, nem minősülnek orvosi szakvéleménynek, nem pótolják az orvosi kivizsgálást és kezelést. A cikk tartalmát a Wikipédia önkéntes szerkesztői alakítják ki, és bármikor módosulhat. |